在 Intel Gaudi 2 上加速蛋白質語言模型 ProtST

簡介
蛋白質語言模型 (PLM) 已成為預測和設計蛋白質結構和功能的強大工具。在 2023 年國際機器學習大會 (ICML) 上,MILA 和 Intel Labs 釋出了 ProtST,這是一種基於文字提示的開創性多模態蛋白質設計語言模型。自那時起,ProtST 在研究界廣受歡迎,在不到一年的時間裡被引用超過 40 次,顯示了該工作的科學實力。
PLM 最流行的任務之一是預測氨基酸序列的亞細胞定位。在此任務中,使用者將氨基酸序列輸入模型,模型輸出一個標籤,指示該序列的亞細胞定位。開箱即用的零樣本 ProtST-ESM-1b 優於最先進的少樣本分類器。
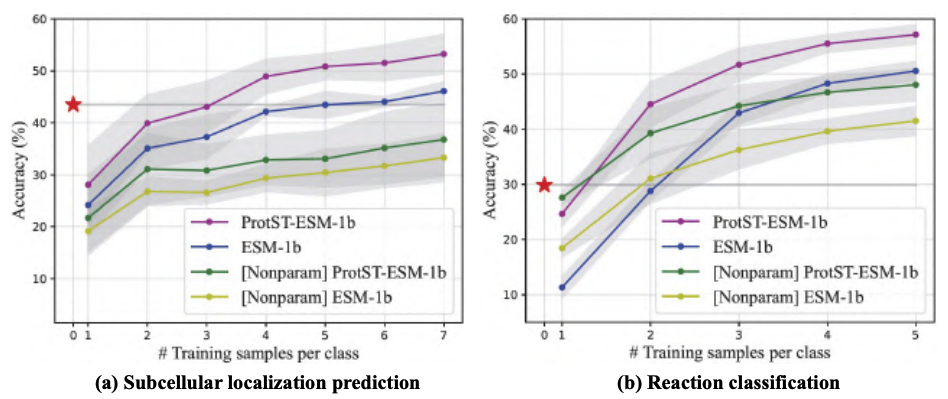
為了使 ProtST 更易於訪問,Intel 和 MILA 重新架構並在 Hugging Face Hub 上共享了該模型。您可以在此處下載模型和資料集。
本文將向您展示如何使用 Intel Gaudi 2 加速器和 Optimum for Intel Gaudi 開源庫高效執行 ProtST 推理並對其進行微調。Intel Gaudi 2 是英特爾設計的第二代 AI 加速器。請檢視我們的上一篇部落格文章,瞭解深入介紹以及透過 Intel Developer Cloud 訪問它的指南。藉助 Optimum for Intel Gaudi 庫,您只需進行最少的程式碼更改即可將基於 transformer 的指令碼移植到 Gaudi 2。
ProtST 推理
常見的亞細胞定位包括細胞核、細胞膜、細胞質、線粒體等,此資料集中對此有更詳細的描述。
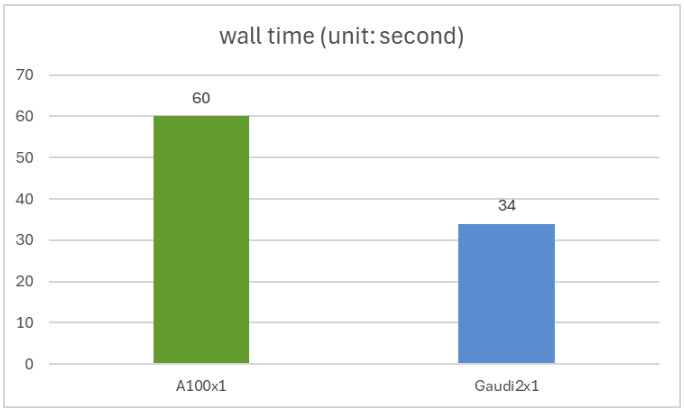
我們使用 ProtST-SubcellularLocalization 資料集的測試集,比較 ProtST 在 NVIDIA A100 80GB PCIe 和 Gaudi 2 加速器上的推理效能。該測試集包含 2772 個氨基酸序列,序列長度從 79 到 1999 不等。
您可以使用此指令碼重現我們的實驗,其中我們以全 bfloat16 精度執行模型,批處理大小為 1。我們在 Nvidia A100 和 Intel Gaudi 2 上獲得了相同的 0.44 精度,Gaudi2 的推理速度比 A100 快 1.76 倍。單個 A100 和單個 Gaudi 2 的實際時間如下圖所示。
ProtST 微調
在下游任務上對 ProtST 模型進行微調是提高建模準確性的一種簡單且成熟的方法。在此實驗中,我們將模型專門用於二元定位,這是亞細胞定位的一個更簡單的版本,其二元標籤指示蛋白質是膜結合還是可溶的。
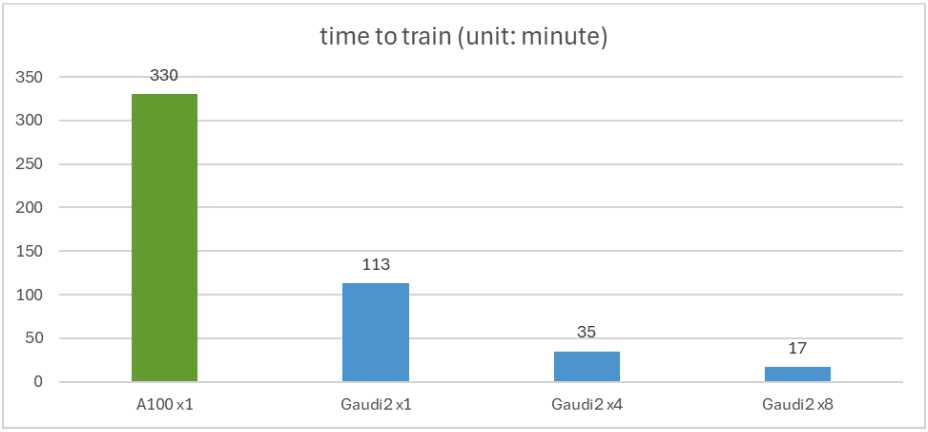
您可以使用此指令碼重現我們的實驗。在這裡,我們以 bfloat16 精度在 ProtST-BinaryLocalization 資料集上對 ProtST-ESM1b-for-sequential-classification 模型進行微調。下表顯示了在不同訓練硬體設定下測試集上的模型準確性,它們與論文中釋出的 MLLM 結果(約 92.5% 的準確性)非常接近。

下圖顯示了微調時間。單個 Gaudi 2 比單個 A100 快 2.92 倍。該圖還顯示了分散式訓練如何透過 4 或 8 個 Gaudi 2 加速器近乎線性地擴充套件。
結論
在這篇部落格文章中,我們展示了在基於 Optimum for Intel Gaudi 加速器的 Gaudi 2 上部署 ProtST 推理和微調的簡易性。此外,我們的結果顯示了與 A100 相比具有競爭力的效能,推理速度提高了 1.76 倍,微調速度提高了 2.92 倍。以下資源將幫助您開始在 Intel Gaudi 2 加速器上使用您的模型:
感謝您的閱讀!我們期待看到您基於 Intel Gaudi 2 加速器功能,在 ProtST 之上構建的創新成果。

